Introduzione
Il mondo della ricerca gira velocemente e con esso il mondo della tecnologia. Mentre da un lato abbiamo scienziati alla ricerca di risultati sempre più rapidamente, dall’altra abbiamo tecnologie in continua evoluzione al fine di soddisfare le richieste dei ricercatori. La necessità di ottenere risultati affidabili e in poco tempo ha spinto numerose aziende di microscopia e non, a sviluppare hardware e software sempre più potenti. Oltre a rendere gli strumenti sempre più performanti e facili da utilizzare, grandi sforzi sono stati impiegati per lo sviluppo di software d’analisi. Infatti, di fronte a strumenti in grado di produrre enormi quantità di dati sempre più complessi, la necessità di velocizzare i processi di analisi diventa fondamentale.
Un brillante esempio di ottimizzazione del processo di acquisizione e di analisi dati è rappresentato da Nanolive, azienda che è riuscita a ridurre ai minimi termini le difficoltà legate agli esperimenti live imaging, sia dal punto di vista hardware che software (Figura 1). Grazie alla sua innovativa tecnologia, gli strumenti Nanolive permettono di acquisire immagini in time-lapse di colture cellulari senza l’utilizzo di marcatori a fluorescenza. Tali microscopi producono video che vengono definiti high-content e multiplexed, ovvero immagini ricche di informazioni, in cui numerose strutture e processi possono essere visualizzate in 3 dimensioni e nel tempo. È dunque evidente la necessità di software altrettanto potenti in grado di estrapolare informazioni e risultati da questi file complessi.
Negli ultimi anni, Nanolive ha rilasciato svariati software di analisi volti a convertire i propri video in risultati quantificabili. In questa Application Note descriveremo tutti i moduli di analisi Nanolive attualmente disponibili, ponendo particolare attenzione ai vantaggi in termini di tempo e dati ottenibili.
L’olotomografia di Nanolive
Nanolive è un microscopio olotomografico in grado di visualizzare cellule vive in 3D e in time-lapse senza l’utilizzo di marcatori fluorescenti. Grazie alla sua tecnologia innovativa, Nanolive riesce ad ottenere una ricostruzione tridimensionale e ad alta risoluzione dell’indice di rifrazione delle cellule, senza alterare il campione. Infatti, eliminando completamente la fluorescenza, anche i problemi legati alla fototossicità vengono rimossi. Nanolive rappresenta dunque un approccio non-invasivo che consente di semplificare notevolmente la preparazione del campione e la configurazione del protocollo: con i microscopi Nanolive è sufficiente piastrare le colture cellulari e il live-cell imaging può iniziare senza ulteriori passaggi. Per ulteriori informazioni, è possibile consultare il nostro sito MSL.
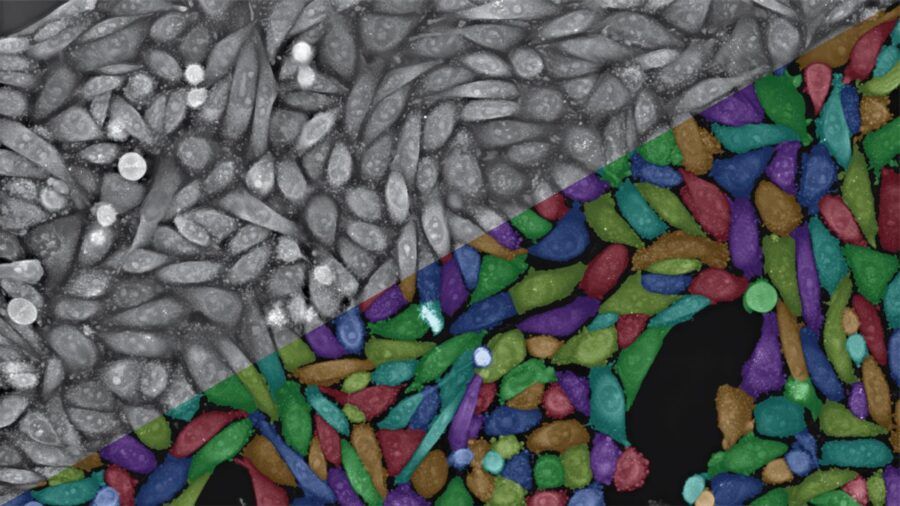
Fig. 1. Immagine ottenuta con Nanolive CX96focus. A sinistra è possibile osservare l’immagine originale in scala di grigi: attraverso l’olotomografia Nanolive è in grado di quantificare direttamente l’indice di rifrazione e generare un’immagine ad alta risoluzione senza l’utilizzo di marcatori. Una volta acquisito il video, il software di analisi Nanolive è in grado di segmentare le cellule e distinguerle tra esse (lato destro dell’immagine). Avvenuto il riconoscimento, i vari moduli di analisi (Digital Assays) forniranno numerosi parametri in pochissimi click.
EVE Analytics: software standard, risultati advanced
Il primo software d’analisi introdotto da Nanolive fu EVE Analytics nel 2021, un modulo base per il riconoscimento e la segmentazione di cellule in video olotomografici. EVE Analytics, anche detto EA, è in grado di distinguere le cellule l’una dall’altra per poi analizzarle utilizzando 13 metriche in parallelo. In pochissimi click, EA fornisce una robusta descrizione del campione a più livelli: popolazione, morfologia e contenuto cellulare. Anche con esperimenti long-term o in esperimenti dove la confluenza cellulare raggiunge livelli significativi, l’algoritmo di EA mostra ottime prestazioni senza riduzioni della qualità della segmentazione.
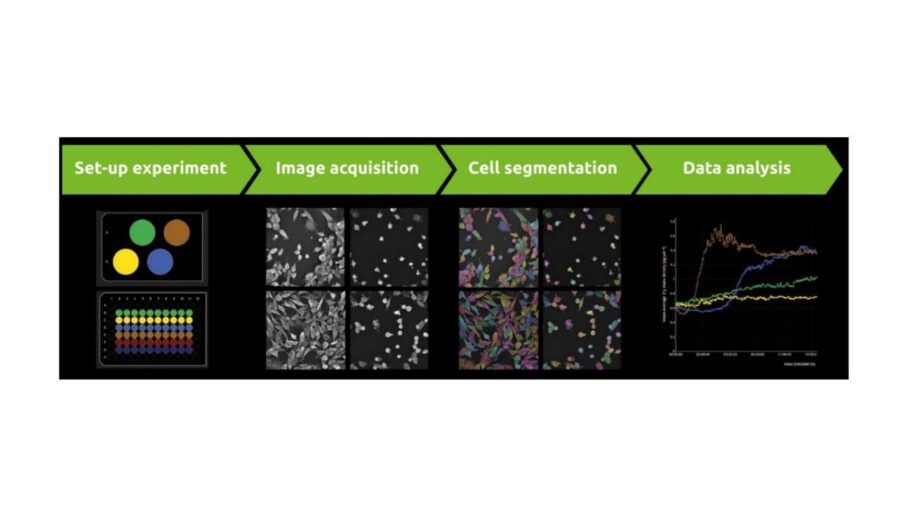
Fig. 2A. Una rappresentazione schematica del processo di acquisizione e analisi con Nanolive. L’esperienza user-friendly sia dal punto di vista hardware che software consente di ottenere numerosi parametri di numerosi campioni in pochissimo tempo.
L’esperienza con EA risulta semplice e immediata: l’operatore è guidato attraverso 2 semplici step e sarà in grado di avviare l’analisi in pochissimi click (Figura 2A). Il primo step consiste in una clusterizzazione dei vari frame del video acquisito, finalizzata all’ottimizzazione dell’analisi. Il secondo step invece consiste nella selezione della miglior treshold (livello di sensibilità) per ogni cluster, e avvierà l’analisi finale. Al termine di questi 2 semplici passaggi, l’operatore otterrà sia le masks (maschere derivanti dalla segmentazione) sia 13 parametri per ogni video. Questi parametri (o metrics) descrivono il campione in maniera dettagliata su 3 livelli: popolazione, morfologia e contenuto (Figura 2B). Mentre per Population e Morphology ritroviamo parametri comuni come numero, confluenza o circolarità, estensione, area, ecc., per Content possiamo ottenere metriche uniche ed esclusive dell’olotomografia. Grazie alla quantificazione diretta dell’indice di rifrazione di Nanolive infatti, lo strumento è in grado di determinare il contenuto cellulare, anche definito come Dry Mass (Massa Secca). La Dry Mass, assieme alla sua distribuzione, definita dal parametro Granularity, sono due metriche che permetto di ottenere importanti informazioni riguardo lo stato del campione o della cellula.
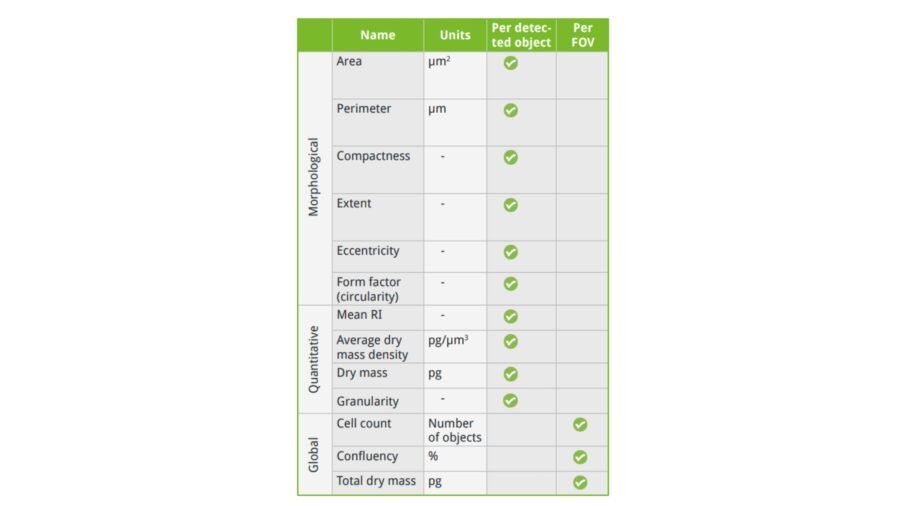
Fig. 2B. Elenco dei 13 parametri del modulo Standard di EA. La dry mass viene calcolata in 3 diverse modalità: dry mass, total dry mass e average dry mass density. Le prime due sono esattamente lo stesso parametro semplicemente calcolato sul singolo detected object (cellula) o sull’intera immagine (popolazione) rispettivamente, mentre average dry mass density è la concentrazione di massa secca per volume di ogni cellula segmentata.
È importante sottolineare che EA è il software sul quale tutti i moduli descritti di seguito si basano. Infatti, le 13 metriche prodotte da EA sono essenziali per i prossimi moduli, i quali andranno ad aggiungere informazioni specifiche oppure ad integrarli con altre.
Live T Cell Assay: magia per l’immuno-oncologia
Il primo modulo d’analisi specializzato introdotto da Nanolive fu il Live T Cell Assay, un software specifico per il settore dell’immuno-oncologia. Una delle strategie più promettenti e diffuse nella ricerca contro il cancro è l’immunoterapia, approccio che si basa sulla capacità di attivare cellule del sistema immunitario come i linfociti T (T Cell) contro le cellule tumorali. Pertanto, analizzare l’abilità di indurre morte cellulare delle T Cell è essenziale per valutare l’efficacia del trattamento. A tal proposito, un esperimento comunemente praticato consiste nel coltivare T Cell insieme a cellule tumorali al fine di osservare le interazioni tra le due popolazioni cellulari e testare la citotossicità delle T Cell. Questi esperimenti, detti co-colture, sono però caratterizzati da numerosi eventi e interazioni tra le due popolazioni nel tempo, per cui l’utilizzo di tecniche di microscopia time-lapse come Nanolive risulta fondamentale.
Data la complessità dell’esperimento anche la sua analisi risulta complessa…e invece no! Grazie al modulo Live T Cell Assay è possibile analizzare tutte le interazioni ed eventi nelle co-colture con un solo click. LTCA è il primo software al mondo in grado di distinguere due popolazioni cellulari in label-free, infatti classifica le cellule immunitarie da quelle tumorali senza necessità di marcatori a fluorescenza (Figura 3A). Una volta classificate le due popolazioni, LTCA integra i 13 parametri di EA e in maniera automatica fornirà il profilo fenotipico di entrambe le popolazioni e la quantificazione di ogni evento avvenuto nel corso della co-coltura. Nello specifico, LTCA segmenta le T Cell (rosso), le cellule tumorali (blu) e le T Cell in contatto con le Target Cell (giallo).
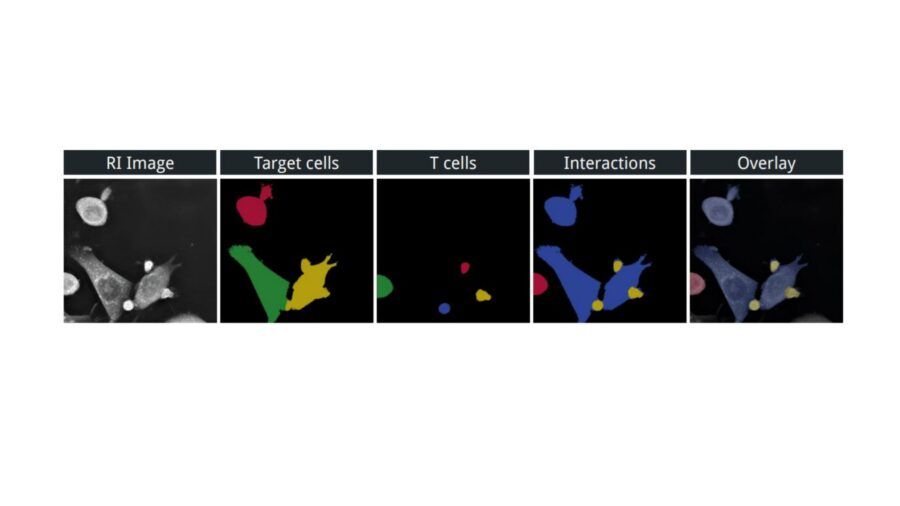
Fig. 3A. Le maschere generate da Eve Analytics. Solo con l’indice di rifrazione (RI) il software è in grado di discriminare le Target Cell, le T Cell e le loro interazioni.
Ottenute le maschere, il software sarà in grado di analizzare e quantificare i 4 differenti step prima menzionati: Find (Figura 3B), Bind (Figura 3C), Stress (Figura 3D) e Kill (Figura 3E).
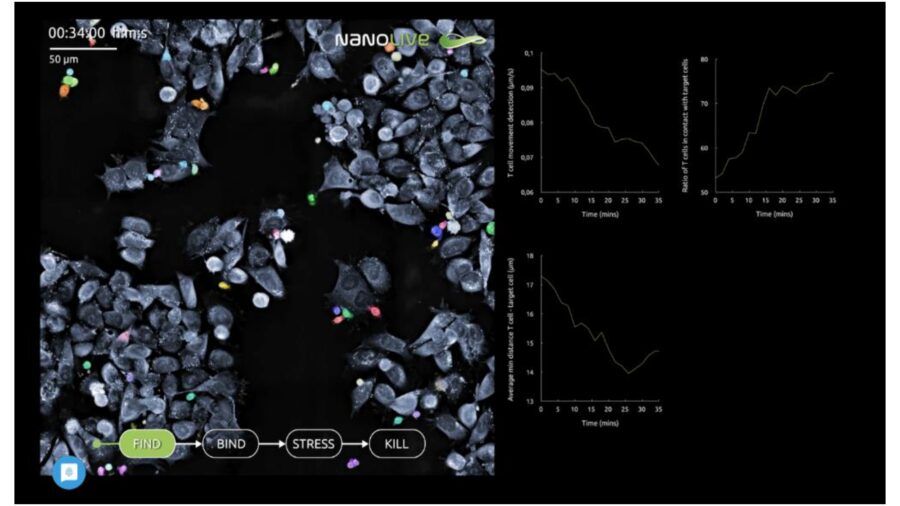
Fig. 3B. Find. Calcolando la motilità delle T Cell, la loro distanza dalle Target Cell e la percentuale di T Cell in contatto con le cellule tumorali Eve Analytics fornisce quantificazione e tempistiche delle T Cell che hanno trovato le Target Cell.
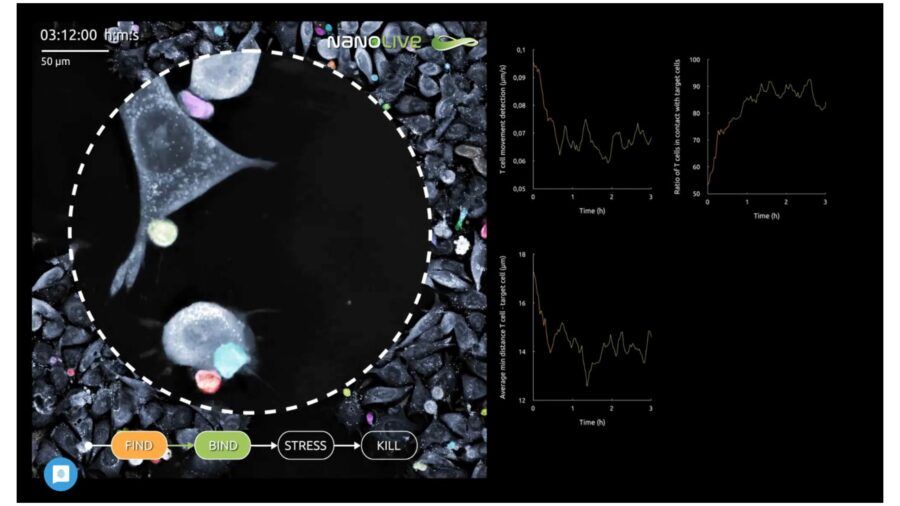
Fig. 3C. Bind. Utilizzando gli stessi parametri, il software rileva anche il numero di T Cell “legate” alle Target Cell. In questo caso i movimenti delle T Cell saranno ridotti a causa del legame e tale rallentamento permette al software di discriminarle.
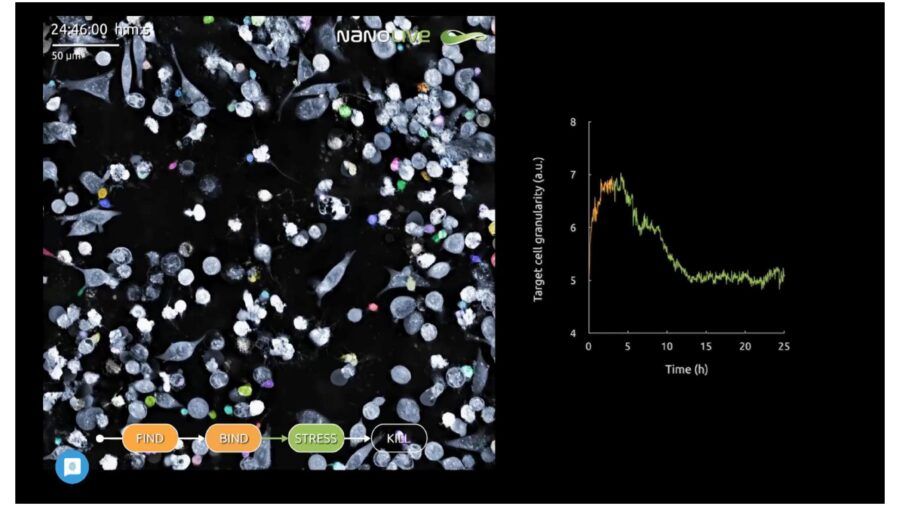
Fig. 3D. Stress. Dopo il Bind, i linfociti T inducono stress nella cellula Target; in questa fase, la quantificazione della Granularity delle Cellule tumorali permette di notare gli effetti di tale stress. Una riduzione della “granularità”, dunque della distribuzione degli organelli e del contenuto cellulare, rappresenta infatti un sintomo di stress.
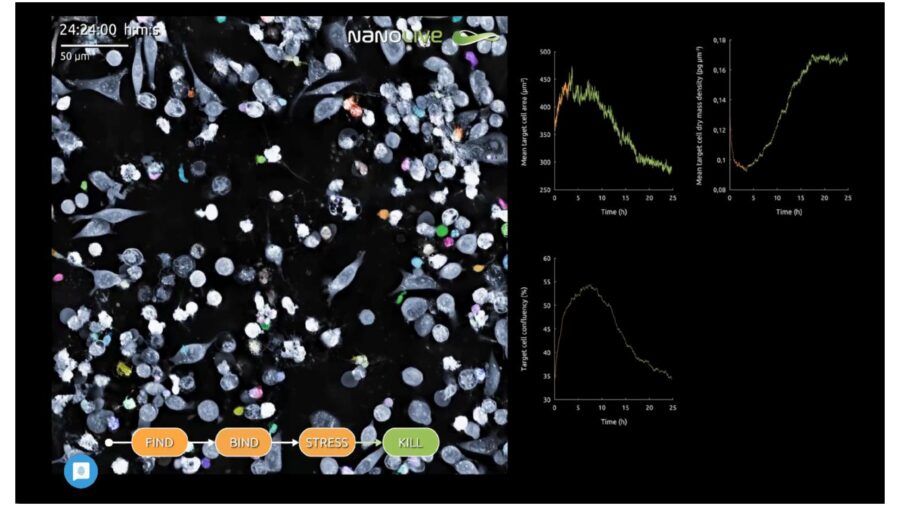
Fig. 3E. Kill. Successivamente i linfociti T causano la morte della cellula tumorale. Combinando la semplice quantificazione della Target Cell Area o Confluency e la quantificazione della Dry Mass (la perdita d’acqua è sintomo di morte cellulare) il software rileva immediatamente la morte delle cellule tumorali.
In conclusione, Nanolive offre una soluzione completa ed efficace per la visualizzazione e l’analisi di co-colture di linfociti e cellule tumorali, in maniera user-friendly e del tutto automatica, per risultati immediati e stupefacenti.
Smart Lipid Droplet AssayLIVE: dimentica la conta manuale
Una delle strutture facilmente riconoscibili nei video olotomografici di Nanolive sono le Lipid Droplet (LD), agglomerati lipidici subcellulari di forma sferica che si trovano in tutte le cellule eucarioti (Figura 4A). Considerate semplici inclusioni citoplasmatiche per decenni, le LD stanno emergendo negli ultimi anni come veri e proprio organelli con funzioni chiave nel metabolismo e nell’omeostasi cellulare. Anche in questo settore, la necessità di analizzare e monitorare nel tempo questi numerosi e picocli organelli subcellulari diventa essenziale. Alla luce di ciò Nanolive ha rilasciato un modulo di analisi dedicato dal nome SLDA: dopo l’acquisizione label-free con SLDA è possibile ottenere una dettagliata analisi di tutte le LD nel corso dell’esperimento.
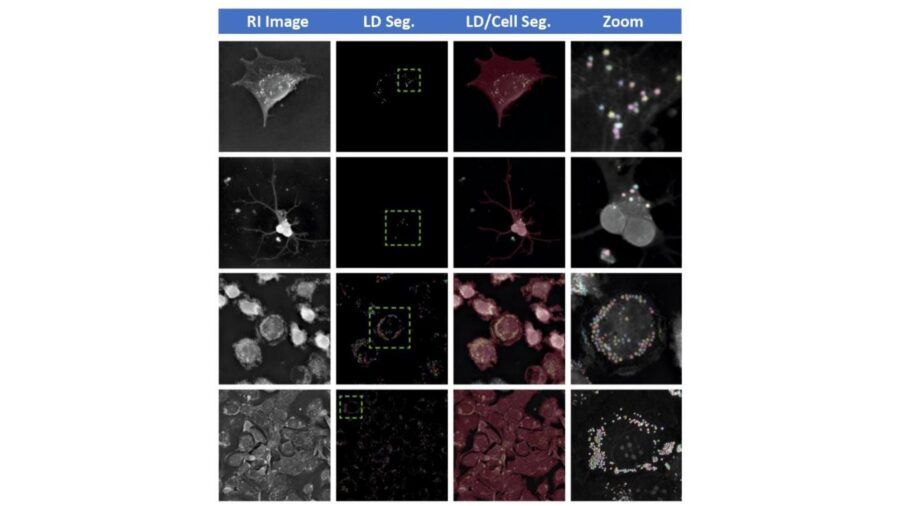
Fig. 4A. Nella stragrande maggioranza delle cellule è possibile osservare la presenza di agglomerati lipidici sferici, le Lipid Droplets. Basandosi esclusivamente sull’indice di rifrazione il software SLDA è in grado di discriminare LD e cellule, generando delle maschere.
Tale Digital Assay permette la segmentazione label-free delle LD con un’accuratezza maggiore rispetto ai sistemi fluorescence-based [1]. Ottenute le maschere di segmentazione (Figura 4A), SLDA calcola 39 parametri garantendo un’analisi completa delle dinamiche delle LD: quantificazione del numero assoluto o per cellula, dei parametri morfologici (forma e dimensione), del contenuto e della distribuzione spaziale all’interno della cellula (Figura 4B). Il tutto, con pochissimi click; per la precisione, gli stessi di EA.
[1] Puoi scaricare la completa e dettagliata Technical Note a questo link: SLDA
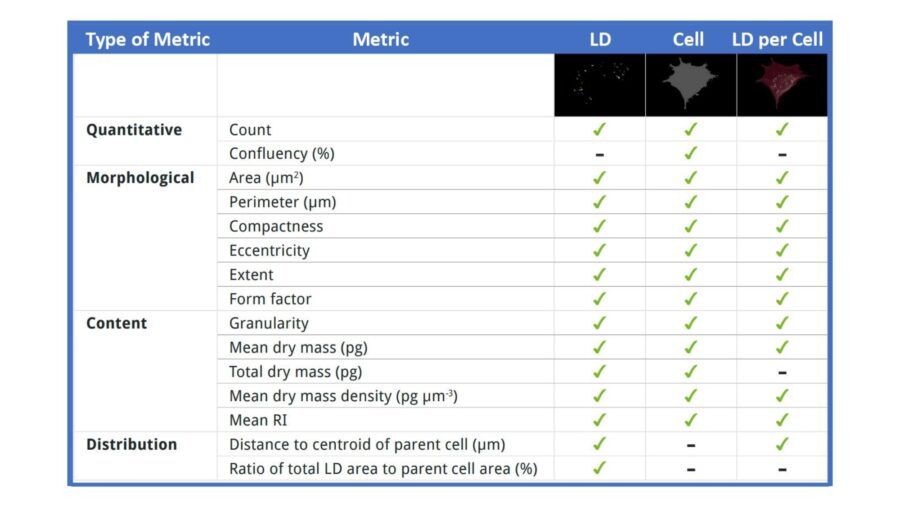
Fig. 4B. Lista di tutti i parametri quantificati da SLDA in maniera automatica. I parametri vengono calcolati per LD, per cellula o per entrambi combinati fornendo un’analisi completa su vari livelli.
Lo Smart Lipid Droplet AssayLIVE è uno strumento digitale unico che attualmente non trova rivali. L’accuratezza e la precisione delle sue analisi, combinate alla tecnologica label-free di Nanolive, offrono una soluzione unbiased e riproducibile per gli studi sul metabolismo cellulare. La possibilità di visualizzare senza fluorescenza LD nel tempo e ad alta frequenza abbatte i limiti tecnologici attuali e apre nuovi orizzonti in numerosi campi di ricerca.
LIVE Cell Death Assay: un click per sapere tutto
I test di vitalità sono esperimenti molto comuni e impiegati per determinare la sopravvivenza e la mortalità di una determinata coltura cellulare. Applicati in vari campi di ricerca, questi test sono spesso utilizzati per misurare lo stato di salute cellulare in risposta a stimoli extracellulari, agenti chimici o trattamenti terapeutici. Infatti, questi esperimenti permettono di effettuare screening di composti al fine di determinarne l’effetto sulla proliferazione, metabolismo o morte cellulare.
Vitalità e mortalità cellulare possono essere individuate e quantificate sulla base di caratteristiche morfologiche, metaboliche o molecolari. Solitamente, le tecniche convenzionali prevedono un approccio semplicistico per definire la vitalità cellulare, andando a quantificare il numero di cellule vive o morte. Questi esperimenti sono spesso end-point assay caratterizzati dall’impiego di staining a fluorescenza, dunque limitati all’analisi di un singolo time-point. Di conseguenza, tali approcci impediscono l’investigazione delle dinamiche della mortalità cellulare nel tempo. Una soluzione a questo limite è rappresentata da esperimenti in time-lapse come l’olotomografia di Nanolive, il quale, non solo consente il monitoraggio della vitalità del tempo, ma non altera il campione e il risultato grazie alla sua tecnologia label-free.
L’imaging unbiased e multiplexed di Nanolive fornisce un’opportunità unica per lo studio della vitalità cellulare sia dal punto di vista dell’imaging, ma anche dell’analisi. Infatti, Nanolive ha sviluppato un software di analisi dedicato ai test di vitalità e mortalità. Il Live Cell Death Assay (LCDA) è la prima soluzione digitale che, con un semplice click, estrapola informazioni complete riguardo vitalità e mortalità cellulare da esperimenti time-lapse interamente label-free. Inoltre, grazie all’impiego dell’intelligenza artificiale, LCDA non solo riconosce e quantifica cellule vive e morte, ma discrimina anche eventi di apoptosi e necrosi. Una volta identificata una cellula morta infatti, LCDA calcola la probabilità che la morte sia avvenuta in maniera programmata (apoptosi) oppure “accidentale” (necrosi) (Figura 5).
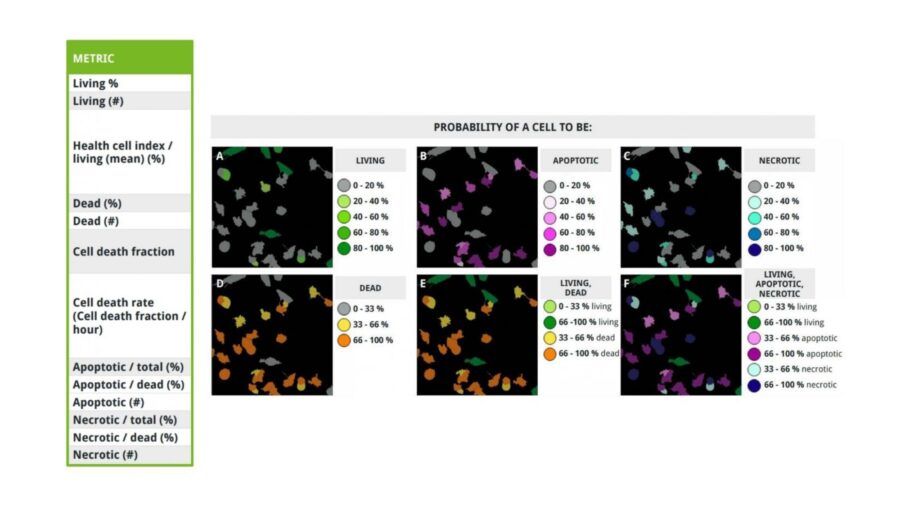
Fig. 5. Overview dei parametri e delle maschere calcolate e prodotte da LCDA. Le cellule vengono segmentate e sfruttando 320 caratteristiche LCDA assegna ogni cellula ad una categoria con una percentuale di confidenza. Ciascuna cellula riceve dunque 3 probabilità (una per categoria) per ogni time-point: la probabilità più alta determina la categoria di appartenenza e dunque il colore con cui è mostrata. Una volta classificate, LCDA quantifica il numero di oggetti per categoria e calcola tutti i parametri elencati.
LCDA è stato allenato con machine learning utilizzando specifici cambiamenti morfologici del citoscheletro per distinguere apoptosi da necrosi, e ulteriori 300 parametri per discriminare cellule vive o morte. Con un solo click, questo Digital Assay segmenta e quantifica numerosi campioni diventando uno strumento estremamente efficace per screening di composti, come riportato in figura 6.
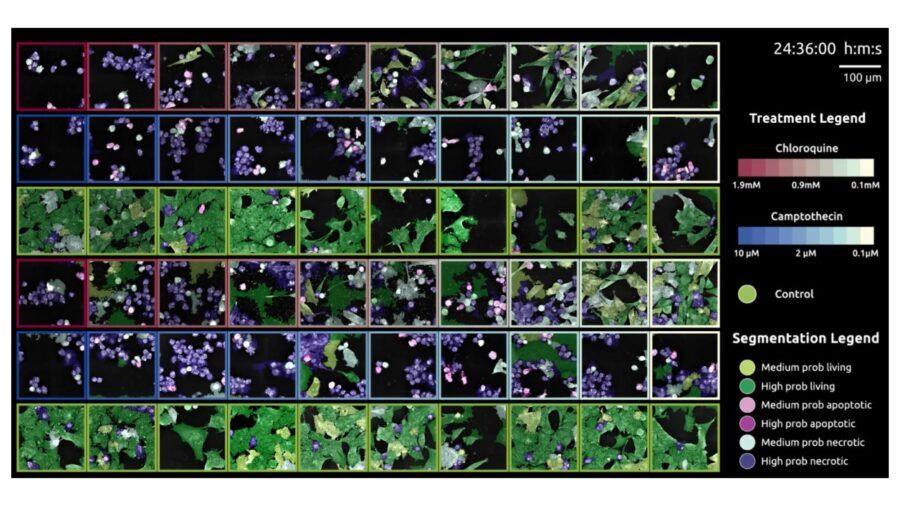
Fig. 6. Esperimento effettuato con Nanolive CX96focus in piastre Multiwell da 96 pozzetti. In questo screening due composti sono stati testati a varie concentrazioni (riportate nella colonna destra). In questa panoramica dei 60 campioni analizzati in parallelo, è possibile notare come LCDA sia in grado di identificare le cellule vive (in verde) da quelle morte (in viola o blu).
LCDA + fluorescenza = LIVE Cytotoxicity Assay
Una delle novità del 2024 è il LIVE Cytotoxicity Assay, il primo Digital Assay di Nanolive in grado di quantificare il segnale a fluorescenza. Nonostante l’olotomografia sia una tecnologia label-free con lo scopo di evitare qualsiasi danno al campione, l’imaging di Nanolive può comunque essere integrato con una sorgente LED per visualizzare staining a fluorescenza. La fluorescenza è utile per moltissime applicazioni, come l’espressione di specifici geni/proteine, strutture subcellulari, sotto-popolazioni, marcatori, composti/nanoparticelle, ecc. Di conseguenza, oltre al segnale olotomografico, anche il segnale a fluorescenza rappresenta un’importante fonte di risultati e la sua quantificazione diventa essenziale.
Il motivo dell’integrazione della fluorescenza con LCDA nasce dalla necessità di moltissimi ricercatori di distinguere differenti popolazioni cellulari all’interno dello stesso pozzetto. In molti test di citotossicità infatti, varie tipologie cellulari vengono testate contemporaneamente al fine di valutare la specificità del trattamento o del composto. Pertanto, monitorare con precisione i diversi effetti dello stesso trattamento sulle differenti sub-popolazioni diventa essenziale. Grazie a LCA, tutto ciò è possibile con pochissimi click. Sfruttando l’imaging label-free e l’intelligenza artificiale di LCDA il software determinerà con precisione cellule vive, apoptotiche e necrotiche, mentre grazie alla nuova funzione potremmo selezionare le differenti popolazioni (opportunamente marcate con fluorescenza), ottenendo così dati specifici per ogni sotto-popolazione lungo tutta la durata dell’esperimento (Figura 7). LCA fornirà un dettagliato report sulla vitalità e mortalità delle sub-populations (chiamate “bio-pops”), ma anche tutti i valori a livello di morfologia e contenuto cellulare, solitamente ottenuti dal modulo di analisi base EVE Analytics.
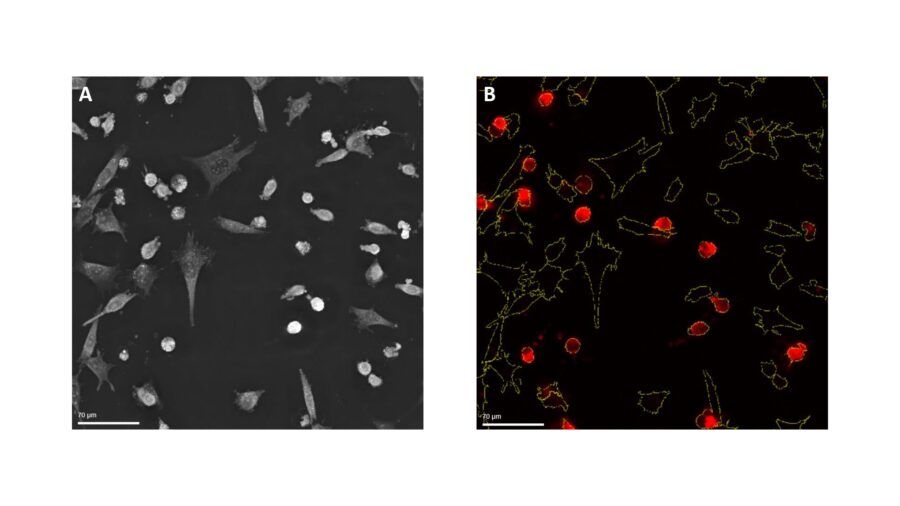
Fig. 7. Immagine olotomografica acquisita con Nanolive CX96focus, nello specifico un singolo frame di un video di 18 ore. Alcune cellule appaiono distese, mentre altre più rotonde. B. In rosso la marcatura fluorescente di una delle due popolazioni. In giallo, le masks (i contorni) delle cellule estrapolate da LCA basandosi sull’immagine olotomografica. Grazie all’integrazione di queste due informazioni (segnale a fluorescenza e maschere) LCA è in grado di riconoscere e distinguere le due popolazioni. In questo esempio, la popolazione “rossa” sta verosimilmente andando incontro a morte cellulare programmata, rispetto alla popolazione “label-free” che appare più in salute.
Tuttavia, l’integrazione della fluorescenza con LCDA non deve far credere che la sua funzione sia limitata alla sola ed esclusiva determinazione di bio-pops. Infatti, la capacità di quantificare il segnale a fluorescenza apre numerose possibilità, come il monitoraggio dell’espressione di un gene e/o proteina nel tempo oppure l’uptake di composti o nanoparticelle. Il software fornirà sempre tutti i parametri disponibili di popolazione, morfologia, contenuto cellulare e vitalità/morte cellulare, ma grazie alla nuova funzione potremo monitorare nel tempo la fluorescenza. Due esempi di queste nuove possibilità sono stati descritti in dettaglio in due nostri articoli tecnici precedentemente pubblicati (LCA2 e LCA3).
Smart Mitochondria Assay: il futuro sta arrivando
Assieme alle Lipid Droplets, i mitocondri sono gli organelli più riconoscibili nei filmati olotomografici di Nanolive. Un software per la loro analisi non è ancora disponibile, ma recentemente è stato annunciato lo Smart Mitochondria Assay (Figura 8). Non sappiamo ancora nulla di questo modulo, ma visti i suoi “cugini” non possiamo che aspettarci il meglio. Segui MS News Lab per aggiornamenti!
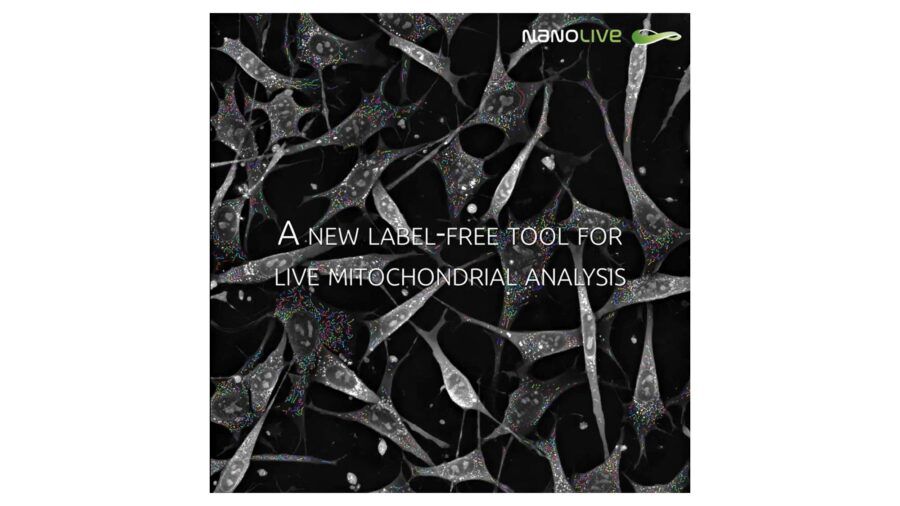
Fig. 8. Prime immagini di Smart Mitochondria Assay nelle quali è possibile visualizzare la segmentazione di ogni singolo mitocondrio.
Conclusione
Ottenere risultati affidabili e riproducibili è l’unico obiettivo che accomuna tutti i ricercatori del mondo. In questa application note abbiamo analizzato come Nanolive abbia contribuito enormemente a soddisfare le esigenze di affidabilità e rapidità con uno strumento perfettamente specializzato dal punto di vista hardware e estremamente potente dal punto di vista software. L’imaging label-free di Nanolive rimuove ogni bias legato alla fototossicità e alla qualità dello staining, mentre i suoi Digital Assay automatizzano e velocizzano analisi complicatissime. Come descritto, i moduli di analisi Nanolive sono estremamente user-friendly e forniscono un’ampia gamma di metriche, sia generali che specifiche/specializzate. A breve, un nuovo Digital Assay dedicato ai mitocondri verrà rilasciato, ma chissà quante altre novità ci attendono nel 2025. La tecnologia e l’Intelligenza artificiale migliorano di giorno in giorno, lasciando presumere l’arrivo di software d’analisi sempre più potenti e sempre più versatili.

